Über die zufällige Entstehung einer nicht reduzierbar komplexen Struktur
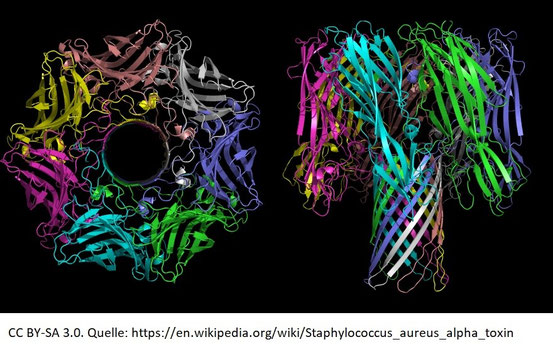
Ein Standard-Argument gegen Evolution seitens Intelligent Design lautet, biologische Systeme seien derart komplex, dass sie nicht in hinreichend kleinen Zwischenschritten evolvieren könnten. Es sei erst recht sehr unwahrscheinlich, dass spezifisch komplexe Strukturen und Funktionen auf einen Schlag entstünden. Dies könne man bereits bei einzelnen Proteinen zeigen, die hochgradig spezifisch für bestimmte Substrate, Wechselwirkungen und Molekularprozesse ausgelegt seien und einen sehr individuellen strukturellen Aufbau zeigten [1] (siehe Abb. 1, rechts, als Beispiel für ein komplexes Membranprotein).
Das Stichwort hierzu lautet nicht reduzierbare Komplexität. Wir zeigen hingegen, dass solche Strukturen (hier: ein komplettes Protein mit einer komplexen Funktion und mehreren spezifischen Bindestellen) bereits in kurzer Zeit de novo evolvieren können. Ein besonders eindrückliches und schönes Beispiel steckt hinter der Geschichte des t-urf13-Proteins, das wir hier vorstellen.
Die Vorgeschichte: Heterosis-Mais
Anfang des 20. Jahrhunderts versuchte man in den USA, robuste Mais-Sorten zu züchten, die auch unter schwierigen Bedingungen gut wachsen. Bald stellte sich heraus, dass insbesondere Heterosis-Pflanzen - mischerbige Pflanzen mit genetisch deutlich unterschiedlichen Eltern - am widerstandsfähigsten sind. Das Problem bei der Anzucht ist jedoch, dass der Heterosis-Grad, also das erwünschte, hohe Ausmaß an Mischerbigkeit, mit jeder nachfolgenden Generation sinkt, sodass der physiologische Vorteil ziemlich bald verschwindet.
Um diesem Effekt zu begegnen, züchtete man in einer der beiden vorgesehenen elterlichen Linien eine Pollensterilität. Daher sind diese Pflanzen auf Fremdbestäubung angewiesen. Somit konnte man Jahr für Jahr wieder "frischen" Heterosis-Saatmais erzeugen. Dies gelang mit verschiedenen Zuchtlinien.
Pollensteriler Mais
Ein Standard-Argument gegen Evolution seitens Intelligent Design lautet, biologische Systeme seien derart komplex, dass sie nicht in hinreichend kleinen Zwischenschritten evolvieren könnten. Es sei erst recht sehr unwahrscheinlich, dass spezifisch komplexe Strukturen und Funktionen auf einen Schlag entstünden. Dies könne man bereits bei einzelnen Proteinen zeigen, die hochgradig spezifisch für bestimmte Substrate, Wechselwirkungen und Molekularprozesse ausgelegt seien und einen sehr individuellen strukturellen Aufbau zeigten[1] (siehe Abb. 1, rechts, als Beispiel für ein komplexes Membranprotein).
Das Stichwort hierzu lautet nicht reduzierbare Komplexität. Wir zeigen hingegen, dass solche Strukturen (hier: ein komplettes Protein mit einer komplexen Funktion und mehreren spezifischen Bindestellen) bereits in kurzer Zeit de novo evolvieren können. Ein besonders eindrückliches und schönes Beispiel steckt hinter der Geschichte des t-urf13-Proteins, das wir hier vorstellen.
Die Vorgeschichte: Heterosis-Mais
Anfang des 20. Jahrhunderts versuchte man in den USA, robuste Mais-Sorten zu züchten, die auch unter schwierigen Bedingungen gut wachsen. Bald stellte sich heraus, dass insbesondere Heterosis-Pflanzen - mischerbige Pflanzen mit genetisch deutlich unterschiedlichen Eltern - am widerstandsfähigsten sind. Das Problem bei der Anzucht ist jedoch, dass der Heterosis-Grad, also das erwünschte, hohe Ausmaß an Mischerbigkeit, mit jeder nachfolgenden Generation sinkt, sodass der physiologische Vorteil ziemlich bald verschwindet.
Um diesem Effekt zu begegnen, züchtete man in einer der beiden vorgesehenen elterlichen Linien eine Pollensterilität. Daher sind diese Pflanzen auf Fremdbestäubung angewiesen. Somit konnte man Jahr für Jahr wieder "frischen" Heterosis-Saatmais erzeugen. Dies gelang mit verschiedenen Zuchtlinien.
Pollensteriler Mais
Viele der pollensterilen Linien wurden genetisch untersucht um herauszufinden, worauf die Pollensterilität beruht. Es war zu erwarten, dass irgendwo im Genom ein Gen ausgefallen war, das für die Pollenreifung essenziell ist. Überraschenderweise erwies sich beim Texas Male-Sterile Maize ein gänzlich anderer Effekt als verantwortlich für die Pollensterilität: Im Genom der Mitochondrien wurde ein komplett neues Gen gefunden, welches für die Sterilität verantwortlich war. Seine Entdecker nannten es t-urf13.
t-urf13: Aufbau und Molekularphysiologie
Weitere Untersuchungen förderten Erstaunliches über t-urf13 zu Tage. Etliche Arbeiten der nächsten Jahre (Veröffentlichungen [2] bis [13]) zeigten, dass es sich um ein mitochondriales Membranprotein handelt. Das bedeutet, es sitzt als Multimer (im Verbund mit mehreren, meist vier bis fünf t-urf-13-Molekülen) in der Membran und fungiert als Kanalprotein. Es bewirkt zu einer definierten Zeit der Blütenreifung gezielt das Absterben der betreffenden Zellen, sodass die gesamte Pflanze völlig intakt und vital bleibt, aber eben keine Pollen bildet. Ferner besitzt das Protein eine spezifische Bindestelle für eine Gruppe von Pilz-Toxinen.
Der dafür verantwortliche Mechanismus des t-urf13-Proteins ist kompliziert und hochspezifisch:
· Es bildet in der Membran multimere Strukturen, die als gesteuerte Ionenkanäle fungieren. Der Komplex kann unterschiedliche Raumstrukturen (Konformationen) annehmen.
· Es spricht auf bestimmte Moleküle an, die den Kanal öffnen oder schließen.
Damit ist T-urf13 faktisch ein ligandengesteuerter Ionenkanal. Das Protein verfügt
über mehrere Bindestellen, die hochspezifisch Liganden binden können. Und es kann sich über spezifische Protein-Protein-Interaktionen multimerisieren und den Kanal ausbilden, der die Membran für
bestimmte Ionen durchlässig macht. Dies leitet letztlich die Pollen-Sterilität ein.
Es kommt noch besser: Dieses hochspezifische Protein entstand in einem Schritt durch zufälliges Verschmelzen von Sequenzen, die niemals ihn ihrer Vorgeschichte für Proteine kodierten! Wir gehen darauf noch näher ein.
Will man den Evolutionsgegnern Glauben schenken, sei derartiges astronomisch unwahrscheinlich. Nach Ansicht des amerikanischen Intelligent-Design-Vertreters Michael Behe (2007, S. 142-147) [1] sei es praktisch unmöglich, dass Proteine mit mehr als zwei spezifischen Bindestellen selbst in erdgeschichtlichen Zeiträumen auf natürlichem Weg evolvieren. Bei der Entstehung von zwei spezifischen Bindestellen, die eine Doppelmutation erfordern würden, sei dies gerade noch im Lauf von Jahrmilliarden möglich. Und dies auch nur einmal unter Aber-Trilliarden Trillionen einzelliger Organismen, keineswegs bei komplexen Mehrzellern, geschweige denn bei hochentwickelten Kulturpflanzen.
Seine Berechnungen, die dies am Beispiel der Chloroquin-Resistenz des Malaria-Erregers Plasmodium falciparum zeigen sollen, sind atemberaubend. Wir müssen hier nicht auf die Details eingehen, denn dass seine Voraussetzungen abstrus sind, beweist unser Beispiel: Innerhalb weniger Jahre entstand in Maisplanzen ein Protein, dessen Zahl an spezifischen Bindestellen die Möglichkeiten der Evolution nach Behes Rechenexempel selbst in kosmischen Zeiträumen hoffnungslos übersteigen würde.
Kurzum: Der Mechanismus ist in hohem Maß nicht reduzierbar komplex und wie erwähnt hinsichtlich Struktur und Mechanismus ligandengesteuerten Ionenkanälen analog.
Herkunft des t-urf13
Der kodierende Bereich für t-urf13 war schnell identifiziert, und seine
DNA-Sequenz wurde ermittelt. Es war zu erwarten, dass das t-urf13 von einem anderen Ionenkanal abstammt und dass durch Mutation(en) lediglich seine Aktivität und Spezifität verändert wurde. Doch
weit gefehlt. Das Ergebnis des BLASTens (Ähnlichkeitssuche in der Sequenzdatenbank) ergab etwas anderes (Abb.2 und 3).
T-urf13 ist eine Chimäre aus zwei Anteilen des Mitochondrien-Chromosoms: (a) einer intergenischen Region (also einem Bereich des Chromosoms, das
nicht transkribiert wird und folglich auch nicht für ein Protein kodiert - gelb) sowie einem Teil des Gens für die 26S-rRNA (grün). Dieses Gen wird in RNA transkribiert, aber nicht translatiert.
Das bedeutet, es kodiert ebenfalls nicht für ein Protein. Es enthält kein proteinkodierendes Leseraster.


Dies war der Kenntnisstand von 1987 (Dewey et al. 1987). Seitdem ist die Datenmenge in den
Sequenzdatenbanken um Größenordnungen gestiegen. Insbesondere liegt mittlerweile eine Vielzahl kompletter Genomsequenzen vor, auch die verschiedener Maisarten und Verwandter. Hat der enorme
Datenbestand etwas an der Erkenntnis von damals geändert?
Die erneute Ähnlichkeitssuche (BLAST am 17.09.2018, durchgeführt vom Autor) bestätigt das damalige Ergebnis: Es gibt keine besseren und spezifischeren Treffer als die beiden benannten. Insbesondere in den Protein-Datenbanken gibt es für alle Anteile des t-urf13-Proteins keine signifikanten Treffer außer dem t-urf13 selbst. Somit ist die Herkunft des t-urf13 eindeutig.
Entstehung des t-urf13: eine ungewöhnliche Ein-Schritt-Evolution
Damit liegt hier einer der seltenen Fälle vor, in denen auf einen Schlag eine spezifisch komplexe Struktur als bloßes Zufallsprodukt entstand: Es gab im Mitochondrien-Genom Doppelstrang-Brüche (was in jeder Zelle immer wieder vorkommt), bei deren Reparatur die beiden Anteile (gelb und grün in Abb.2 und 3) fusioniert wurden.
Bei der Reparatur nicht zusammenpassender Enden (über die Reparatur-Mechanismen non-homologous end joining oder single strand annealing) kommt es oft dazu, dass sich die Sequenz an der Verknüpfungsstelle ziemlich regellos verändert. Dadurch erklärt sich das Zustandekommen des "roten Bereichs" in Abb. 2 und 3, für den keine Ähnlichkeiten in den Datenbanken zu finden sind.
Warum der Vergleich von Lebewesen mit Design hinkt
Dieser Vorgang lässt sich mit einem Beispiel verdeutlichen: Stellen Sie sich vor, Sie knipsen
Bilder mit Ihrer Digitalkamera und kopieren sie auf Ihren PC. Dabei werden die Dateien beschädigt, wodurch Anteile neu kombiniert und etliche Bits verändert werden. Es entsteht eine neue,
kleinere Datei, die nicht mehr als Bilddatei lesbar ist. Stattdessen ist nun eine Programmdatei entstanden, die Musikdateien lesen und abspielen kann!
Jeder, der ein wenig Ahnung von Programmierung hat, weiß, dass so etwas nie und nimmer vorkommen kann. Im Fall der Evolution des Proteins T-urf13 ist aber genau dies passiert. Was lernen wir daraus?
- Technische und biologische Systeme sind nur sehr begrenzt vergleichbar.
- Im Gegensatz zur Technik ist eine rein zufällige Entstehung von funktionalen, spezifisch komplexen Strukturen möglich, zumeist in kleinen und kleineren Schritten, gelegentlich – wie bei t-urf13 – auch mal in einem größeren Sprung.
- Intuition ist bei der Betrachtung der Natur allzu oft kein guter Ratgeber.
Mögliche Gegenargumente
Aus der
ID-Perspektive kann man mit nachstehenden Standard-Einwänden rechnen, die noch kurz abgehandelt seien.
1. Homologie bzw. Sequenzähnlichkeiten liefern keinen Entstehungsmechanismus. Niemand hat valide bewiesen, dass t-urf13 durch rekombinante Evolutionsereignisse entstanden ist.
(Züchtungs-)
Experimente sind doch genau das, was ID-Proponenten immer gefordert haben! Also was erwarten sie noch - eine elektronenmikroskopische Filmaufnahme darüber, wie die Gene miteinander verschmolzen
sind? Richtig ist: In den empirischen Wissenschaften gibt es keine Beweise im streng logischen Sinn. Ein trockenes Flussbett ist kein Beweis, dass dort je Wasser floss: Vielleicht hat ja
jemand das "Flussbett" gebaut? Die Strahlungsspektren der Sonne sind kein Beweis für die Kernverschmelzung in ihrem Inneren: Vielleicht will sie nur in diesen schönen Spektren strahlen? Faser-,
Blut- und DNA-Spuren nebst Fingerabdrücken eines Verdächtigen plus seinen Bildern auf der Überwachungskamera sind kein Beweis, dass der Betreffende am Tatort war. Vielleicht hat jemand
diese Spuren gelegt, oder der Täter war ein Doppelgänger mit zufällig identischem Genom. Aber so funktioniert empirische Wissenschaft nicht.
Entscheidend ist, dass t-urf13 vor den Züchtungsexperimenten unbekannt war. Es kommt exklusiv im CMT-Mais vor, entstand also im Rahmen der Züchtung. Zudem kommen
keine anderen Quellen für t-urf13 infrage als die beiden genannten mitochondrialen DNA-Sequenzen. Wenn das Gen nicht vom Himmel gefallen sein soll (und dabei seltsamerweise hochgradig
ähnlich bereits vorhandenen Fragmenten des Mitochondrien-Genoms), kommen wir nicht umhin zu konstatieren, dass es sich aus den vorhandenen Ressourcen (de novo) gebildet hat.
Genrekombinationen sind seit Jahrzehnten bekannt, die enzymatischen Prozesse, die der Rekombination und der Entstehung von t-urf13 zugrunde liegen, gut verstanden.
Kurz: Wir haben einen empirisch wohlbegründeten, sehr gut verstandenen Mechanismus für die Entstehung des t-urf13-Gens vorliegen. Hier ist in jüngster Zeit
direkt im Rahmen der Züchtung eine komplexe Struktur entstanden, deren Bildungsweg wir detailliert nachvollziehen können - fast bis auf das letzte Nukleotid. Also kann man fragen, was
ID-Vertreter hier überhaupt als valides Argument akzeptieren würden.
2. Die Herkunft von t-urf13 ist ungeklärt. Wahrscheinlich stammt es von einem anderen ("intelligent programmierten") Vorläufer ab, der nur minimal mutieren musste, damit das beobachtete Ergebnis heraus kommt.
Wie dargelegt, ist die Herkunft sehr wohl geklärt. Bei der Mächtigkeit der heutigen DNA-Datenbanken besteht kein Zweifel mehr daran, dass die Entstehung
verlief, wie oben geschildert. Es gibt kein Vorläufer-Gen für t-urf13. Wer anderer Meinung ist, möge dies belegen und ggf. wissenschaftlich publizieren.
3. Die DNA-Elemente, aus denen t-urf13 zusammengesetzt ist, haben vom Designer / Schöpfer eine "versteckte Protein-Kodierungskapazität" mitbekommen.
Erstens ist solch ein Argument grundsätzlich unüberprüfbar, denn es kann niemand definieren, was eine solche "versteckte Entwicklungs- oder Kodierungskapazität"
sein soll. Sie wird nur im Nachhinein postuliert. Ergo kann der Kreationist am Ende immer diesen "Joker" ziehen, ohne befürchten zu müssen, widerlegt zu werden.
Zweitens passen die Fakten nicht zu einem solchen Szenario: Vergleicht man die kodierenden Bereiche (open reading frames oder kurz orfs)
von poteinkodierenden Genen verschiedener Spezies, so findet man immer wieder sogenannte InDels (= Einschübe oder Auslassungen) von 3, 6, 9 usw. Basen, also ganzzahligen
Vielfachen von drei. Vergleicht man hingegen die kodierenden Bereich von Genen, die für funktionale RNA-Moleküle kodieren, wie insbesondere die ribosomalen RNAs und erst recht intergenische
Regionen, so findet man InDels der verschiedensten Längen ohne diese "3er-Regel". Die evolutionsbiologische Erklärung hierfür ist denkbar einfach: Kleine und kleinere Einschübe sowie Auslassungen
haben sowohl in ribosomalen RNAs als auch in Proteinen (und erst recht in intergenischen Regionen) an vielen Stellen keine oder nur geringe Auswirkungen. Wenn es jedoch in einem orf zu einem
InDel von 1, 2, 4, 5, 7, 8, 10 usw. Basen kommt, verschiebt sich das Leseraster. Ab dem betroffenen Punkt ist das Protein komplett verändert – und verliert dadurch fast immer seine biologische
Funktion. Infolgedessen werden solche Mutationen negativ ausselektiert.
Die Tatsache, dass rRNAs nicht der "3er-Regel" unterliegen, spricht für sich. Und in der Tat gibt es keinen Hinweis darauf, dass rRNAs kodierend sind oder es je
waren, was für intergenische Regionen erst recht gilt.
4. Die Funktion von t-urf13 ist destruktiv, nicht konstruktiv.
"Na und?", möchte man fragen. Hier wird der biologische Kontext mit seiner Entstehung verwechselt. Was zählt, ist die Tatsache, dass das t-urf13-Protein einen
spezifischen Aufbau hat, spezifische Bindestellen für Liganden und eine spezifische molekulare Funktion. Ob etwas natürlich ist (fruchtbarer Boden oder Wüste) oder intelligent designt
(Medikamente oder Bomben) hat mit der Frage, ob dieses Etwas "konstruktiv" und "gut" ist oder "destruktiv" und "schlecht" nichts zu tun.
5. Zur Entstehung des t-urf13 auf dem Mitochonrien-Genom waren mehrere Rekombinationsereignisse nötig, was das Szenario noch unwahrscheinlicher macht.
Stimmt. Dennoch hat es stattgefunden! Wenn also, wie bereits gesagt, das Gen nicht durch ein Wunder entstanden sein soll, lautet der
Schluss auf die beste Erklärung nach wie vor, dass es durch eine Reihe von Gen-Rekombinationen entstand. Wie erläutert, sind die dafür erforderlichen Prozesse bekannt und mechanismisch sehr
gut verstanden. Oder wollen uns die ID-Vertreter ernsthaft Glauben machen, dass eine versteckte intelligente Macht Anfang des 20. Jahrhunderts den Züchtern "in die Karten spielte"? Das ist
kein Schluss auf die beste Erklärung, das ist Obskurantismus.
6. Der transkribierte Teil von t-urf 13, der den gesteuerten Ionenkanal liefert, ist nur ein kleiner Teil des "de novo" -Gens. Außerdem ist ein Teil des mitochondrialen atp6 beteiligt, und das ist proteinkodierend.
Das atp6 lieferte lediglich den Promotor für t-urf13, und dieser wird und wurde zuvor genauso wenig translatiert wie alle anderen Anteile des
t-urf13-Gens. Es bleibt also dabei: Das komplexe T-urf13-Protein entstand durch wenige Rekombinationsereignisse (und damit faktisch auf einen Schlag, weil "positiv selektierte
Zwischenschritte" nicht existierten).
7. Es gibt eine lebhafte Debatte in den USA um t-urf13. ID-Vertreter haben gezeigt, dass die Argumentation nicht sticht.
Die wesentlichen Einwände haben wir oben abgehandelt. Zudem wird die "kontroverse Diskussion" nur in Blogs und auf christlichen Internet-Seiten
geführt, nicht in der Fachwelt. Die ID-Vertreter sind somit aufgefordert, ihre wissenschaftlichen Daten und Argumente, sofern sie welche haben, in wissenschaftlichen Journals zu
publizieren, dann diskutieren wir weiter.
Literatur
[1] Behe M (2007) The edge of evolution. Free Press, New
York.
[2] Stamper SE, Dewey RE, Bland MM, Levings CS 3rd (1987) Characterization of the gene urf13-T and an unidentified reading frame, ORF 25, in maize
and tobacco mitochondria. Curr Genet. 12(6): 457-63.
[3] Dewey RE, Timothy DH, Levings CS (1987) A mitochondrial protein associated with cytoplasmic male sterility in the T cytoplasm of maize. Proc
Natl Acad Sci U S A. 84(15):5374-8.
[4] Braun CJ, Siedow JN, Williams ME, Levings CS 3rd (1989) Mutations in the maize mitochondrial T-urf13 gene eliminate sensitivity to a fungal
pathotoxin. Proc Natl Acad Sci U S A. 86(12): 4435-9.
[5] Braun CJ, Siedow JN, Levings CS 3rd (1990) Fungal toxins bind to the URF13 protein in maize mitochondria and Escherichia coli. Plant Cell.
2(2):153-61.
[6] Levings CS 3rd. 1990 The Texas cytoplasm of maize: cytoplasmic male sterility and disease susceptibility. Science
250(4983):942-7.
[7] Korth KL, Kaspi CI, Siedow JN, Levings CS 3rd (1991) URF13, a maize mitochondrial pore-forming protein, is oligomeric and has a mixed
orientation in Escherichia coli plasma membranes. Proc Natl Acad Sci U S A. 88(23):10865-9.
[8] Levings CS 3rd, Siedow JN (1992) Molecular basis of disease susceptibility in the Texas cytoplasm of maize. Plant Mol Biol.
19(1):135-47.
[9] Kaspi CI, Siedow JN (1993) Cross-linking of the cms-T maize mitochondrial pore-forming protein URF13 by N,N'-dicyclohexylcarbodiimide and its
effect on URF13 sensitivity to fungal toxins. J Biol Chem. 268(8):5828-33.
[10] Korth KL, Levings CS 3rd (1993) Baculovirus expression of the maize mitochondrial protein URF13 confers insecticidal activity in cell
cultures and larvae. Proc Natl Acad Sci U S A. 90(8):3388-92.
[11] Rhoads DM, Kaspi CI, Levings CS 3rd, Siedow JN (1994) N,N'-dicyclohexylcarbodiimide cross-linking suggests a central core of helices II in
oligomers of URF13, the pore-forming T-toxin receptor of cms-T maize mitochondria. Proc Natl Acad Sci U S A. 91(17):8253-7.
[12] Siedow JN, Rhoads DM, Ward GC, Levings CS 3rd (1995) The relationship between the mitochondrial gene T-urf13 and fungal pathotoxin
sensitivity in maize. Biochim Biophys Acta. 1271(1):235-40.
[13] Rhoads DM, Levings CS 3rd, Siedow JN (1995) URF13, a ligand-gated, pore-forming receptor for T-toxin in the inner membrane of cms-T
mitochondria. J Bioenerg Biomembr. 27(4):437-45.
Gastbeitrag von: Martin Neukamm
zum vorherigen Blogeintrag zum nächsten Blogeintrag
Liste aller Blogbeiträge zum Thema "Entwicklung allen Seins"
 Philoclopedia
Philoclopedia
Kommentar schreiben